Relações Entre Estrutura e Propriedade/Reatividade/Atividade
Biológica
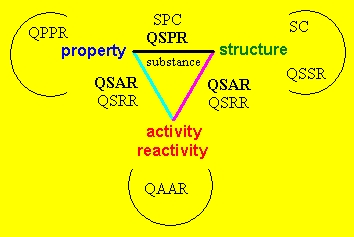
Schematic representation of relationships between
structure, property and activity/reactivity:
QSAR - Quantitative Structure-Activity Relationships
QSPR - Quantitative Structure-Property Relationships
QAAR - Quantitative Activity-Activity Relationships
QPPR - Quantitative Property-Property Relationships
QSSR - Quantitative Structure-Structure Relationships
SPC - Structure-Property Correlation
SC - Structure Correlation
Disciplina
de pós-graduação no 1o semestre de 2003,
QP-399:
Tópicos
Especiais em Físico-Química IX:
“Relações
entre Estrutura e Propriedade/Reatividade/Atividade Biológica”
pelos Profa. Dra. Márcia M. C. Ferreira e Dr.
Rudolf Kiralj,
Instituto de Química, UNICAMP. Horário:
3a. e 5a., 19h às 21h.
Contato: marcia@iqm.unicamp.br,
rudolf@iqm.unicamp.br
EMENTA
Análise Multivariada
Introdução.
Análise exploratória dos dados:
PCA – análise de componentes principais.
HCA – análise hierárquica de agrupamentos.
Construção de modelos de regressão:
PCR – regressão por componentes principais.
PLS – regressão por mínimos quadrados parciais.
Construção de modelos de classificação
(reconhecimento de padrões):
KNN,
SIMCA.
Química Estrutural e Teórica
Introdução.
Parâmetros estruturais de átomos, moléculas
pequenas e macromoléculas.
Análise conformacional estrutural.
Simetria molecular e estereoquímica.
Ligações de hidrogênio.
Interações intra- e intermoleculares.
Complexos moleculares.
Simetria cristalina.
Empacotamento cristalino.
Banco de dados cristográficos.
Breve revisão das teorias aplicadas em química
teórica, computacional e estrutural.
Introdução à teoria dos grafos.
Uso de dados estruturais em química teórica.
Análise conformacional computacional.
Otimização de geometria de complexos
moleculares.
QSAR/QSPR
Introdução.
Quantidades em (Q)SAR/(Q)SPR.
Descritores moleculares globais e locais.
Descritores de complexos moleculares e de empacotamento
cristalino.
Descritores de reatividade e propriedades magnéticas.
Metodologia de (Q)SAR/(Q)SPR.
QSAR-3D.
Técnicas de visualização de moléculas,
complexos moleculares, densidade eletrônica e empacotamento cristalino.
Molecular graphics a nível qualitativo e quantitativo.
Princípios básicos de modelagem molecular.
Docking.
Farmacóforos.
ALEM DE AULAS TEÓRICAS, O CURSO INCLUI
AULAS PRÁTICAS, EXERCÍCIOS, APRESENTAÇÕES DE
TRABALHOS DA LITERATURA E PROJETOS PRÓPRIOS.
Material de curso para fazer download
Notas de Aulas 4, 16-28 são
disponíveis em formato .pdf pequeno (2 páginas originais
numa só) no link português, e formato .pdf grande (1 página
original numa página de pdf) no link inglês. Escolha a opção
melhor para você!
As Notas de Aulas, Exercícios
e os Suplementos são o propriedade de Laboratório de Quimiometria
Teórica e Aplicada, Instituto de Química, Universidade Estadual
de Campinas, Campinas, SP, e portanto se podem usar somente para ensino
e atividade didáticas/educativas (aprendizagem) com citação
adequada! É proibído usar este material ou idéas apresentadas
para fazer qualquer trabalho, escrever textos e fazer qualquer outra coisa
onde o conteudo é modificado e/ou autoria nomeada diferente do que
nestes documentos originais!/
NOVO!
AVISO: Após da data oficial
do fim do semestre, o material do curso (as Notas de Aulas, Exercícios
e Suplementos) não são mais disponíveis para fazer
download. Quem precisa algum material, por favor entrar em contato com
Profa.
Márcia ou Rudolf./
--------------------------------------------------------------------------------------------------------------------------------------------------------------------
–EMENTA (em português)
–Aula No.
1 (18 de fevereiro de 2003): Introdução ao Curso (em português)
–Aula No. 2 (20 de fevereiro de
2003): Introdução à Análise Multivariada. Pré-Processamento
dos Dados (em português)
–Aulas No. 3, 5, 6 e 7 (25 de
fevereiro, 11, 13 e 18 de março de 2003): Análise Exploratória,
partes I, II, III e IV (em português)
–Aula No. 4 (27 de fevereiro de
2003): Introdução à Química Estrutural. Propriedades
Atômicas (em inglês)
–Aulas No. 8, 9, 10 e 11 (20,
25 e 27 de março, e 1 de abril de 2003): Modelos de Regressão,
partes I, II, III e IV (em português)
–Exercícios I/ Exercises
I:Lista
dos Exercícisos I (em
português)/List of Exercises I (in
portugese)Dados
I/Data I
–Aula No. 12 (3 de abril de 2003): Interpretação
de trabalho da literatura, da escholha livre, da área de quimiometria
aplicada - 6 apresentações pelos estudantes (em português)
–Aula No. 13, 14 e 15 (8,
10 e 15 de abril de 2003): Modelos de Classificação (Reconhecimento
de Padrões), partes I, II, III e IV (em português); Interpretação
de trabalho da literatura, da escholha livre, da área de quimiometria
aplicada - 3 apresentações pelos estudantes (8 de abril de
2003; em português)
–Exercícios II:Lista
dos Exercícisos II (em
português)Dados
II
–Aula No. 16 (22 de abril de 2003):
Ligação Química. Comprimento e Ângulo de Ligação,
Ângulo de Torsão (em português)
–Aulas No. 17 e 18 (24 e 29 de
abril de 2003): Outros Parâmetros Estruturais (Geomêtricos)
das Moléculas Pequenas. Análise Conformacional Estrutural
(em inglês)
–Aula No. 19 (6 de maio de 2003):
Outros Parâmetros Estruturais (Geomêtricos) de Macromoléculas.
Isomeria e Configuração e Conformação Absoluta
(em inglês)
–Aula No. 20 (8 de maio de 2003):
Grupos Pontuais e Simetria. Simetria Molecular (em inglês)
–Aula No. 21 (13 de maio de 2003):
Ligações de Hidrogênio e Outras Interações
Intra- e Intermoleculares. Complexos Moleculares (em inglês)
–Aula No. 22 (15 de maio de 2003):
Sistemas Cristalinos. Simetria Cristalina e Grupos Espaciais. Empacotamento
Cristalino I (em inglês) - [É preciso receber uma Xerox
copia do Suplemento no começo da aula]
–Aula No. 23 (20 de maio de 2003):
Empacotamento Cristalino II. Bancos de Dados Cristalográficos e
Outros Bancos de Dados Úteis (em inglês)
-Palestra sobre Cambridge Structrural
Database
–Aula No. 24 (22 de maio de 2003):
Tópicos Especiais da Química Teórica, Computacional
e Estrutural I (em inglês)
Um brevíssimo
manual e um exemplo de output para o programa
PLATON
–Exercícios III:Lista
dos Exercícisos III (em
português)
Esta lista de exercícios
deve ser entregue IMPRETERIVELMENTE até o final da primeira semana
de JUNHO!
A lista pode ser feita utilizando
a versão DEMO de Pirouette./
–Exercícios IV:Lista
dos Exercícisos IV (em
inglês)Dados
III
Esta lista de exercícios
deve ser entregue até o dia 10 de JUNHO! Após nesta data
não vai ser possível entregar a lista. A lista pode ser feita
utilizando programas PLATON com Raswin, e Piroutte./
–Aula No. 25 (3 de junho de 2003):
Tópicos Especiais da Química Teórica, Computacional
e Estrutural II (em inglês) - [É recomendavel fazer download
do Suplementos
A,
B,
C,
D
e E antes de começo da aula]
–Aula No. 26 (5 de junho de 2003):
Introdução à Química Medicinal. Descritores
Moleculares (em inglês) - [É recomendavel fazer download
das seguintes Notas de Aulas sobre QSAR pelo H.
Kubinyi antes de começo da aula: Ligand-Receptor
Interactions, QSAR
Parameters e Hansch
and Free-Wilson Analyses.]
O prazo para entregar a Lista
dos Exercícios IV é prorrogado até o dia 12 de junho.
Visite este site para ver se tiver mais mudanças do prazo!/
–Aula No. 27 (10 de junho de 2003):
Descritores moleculares II. Similaridade Química e Atividade Biológica.
Introdução à Química Medicinal II. (em inglês)
- [É recomendavel fazer download das seguintes Notas de Aulas sobre
QSAR pelo H. Kubinyi antes
de começo da aula: Similaridade
Química e Atividade Biológica, Descoberta
de Fármacos e QSAR
Não Linear.]
O prazo para entregar a Lista
dos Exercícios IV é mais uma vez prorrogado até o
dia 17 de junho!/
–Aula No. 28 (12 de junho de 2003):
Metodologia de QSAR/QSPR. Exemplos de QSAR e QSPR. QSAR-3D (em inglês)
- [É recomendavel fazer download das seguintes Notas de Aulas sobre
QSAR pelo
H. Kubinyi antes
de começo da aula: QSAR
3D, Análise
de Farmacóforos and Virtual
Screening. Também fazer download de pequenos suplementos: análise
de farmacóforos pelo Catalyst, QSAR com
COMFA, COMFA avancada e virtual
screening.] Exemplos de QSAR/QSPR: inibidores de
protease de HIV-1, propriedades físico-químicas
de PAHs, derivados da omeprazola e logP
de PCBs.
–Aula No. 29 (17 de junho de 2003):
Gráficos Moleculares. Modelagem Molecular. Desenho de Fármacos.
(em ingles) - [É recomendavel fazer download das seguintes Notas
de Aulas sobre QSAR pelo H.
Kubinyi antes de começo da aula: Desenho
de Fármacos Através da Estrutura e Desenho
de Fármacos Usando Computadores, Desenho
Combinatorial de Fármacos.] Entrega da
Lista dos Exercícios IV.
--------------------------------------------------------------------------------------------------------------------------------------------------------------------
-Aula No. 30 (26 de junho de 2003): Apresentação
dos Projetos I
-Aula No. 31 (3 de julho de 2003): Apresentação
dos Projetos II. Comentários sobre as Listas de Exercícios.
Mais do que uma aula prática foi dada pela Profa.
Márcia fora do horário oficial, para ensinar os estudantes
como fazer análise quimiométrica com dados concretos. Com
isso, número total das aúlas é maior de 31./
AVISO: Quem quer participar no
cursinho sobre o uso de Cambridge Structural Database em agosto ou setembro,
por favor mandar um e-mail com nome e e-mail completo para Profa.
Márcia ou Rudolf,
para nos teremos possibilidade de contactá-lo na hora certa!/
--------------------------------------------------------------------------------------------------------------------------------------------------------------------
Notas
Materiais Didáticos Relacionados ao Curso
Links às publicações
da Profa. Márcia M. C. Ferreira
Quimiometria Teórica e Aplicada:
Publicações No. / Publications
No. 14, 15,
17,
19,
20,
21,
22,
23,
24,
25,
26,
27,28,
29,
31,
32,
34,
39,
43,
44,
45,
46,
49,
55,
56,
59,
61,
62
SAR/QSAR, SPR/QSPR, SSR/QSSR:
Publicações No. / Publications
No. 16,
30,
33,
35,
36,
37,
38,
40,
41,
42,
47,
48,
50,
51,
52,
53,
54,
57,
58,
60
Gráficos e Modelagem Molecular:
Publicações No. / Publications
No. 13,
18,
35,
36,
38,
51,
52,
53,
57
Links externos (internet)
Molecular Descriptors and QSAR Web News, Chemometrics Web News: http://www.disat.unimib.it/chm/WebMagazine.htm,
http://www.disat.unimib.it/chm/Chemometrics.htm
- University of Milan, Italy
Computational Chemistry List (CCL): http://server.ccl.net/
- USA
The QSAR and Modelling Society - Resources: http://www.ndsu.nodak.edu/qsar_soc/resource.htm
- USA
Homepage of Chemometrics: http://www.acc.umu.se/~tnkjtg/chemometrics/
- Umeå University, Sweden
Tutorials on Chemometrics: http://www.acc.umu.se/%7Etnkjtg/chemometrics/tutorials.html
Chemometrics World - Spectroscopynow.com: http://www.spectroscopynow.com/Spy/basehtml/SpyH/1,1657,2-0-0-0-0-home-0-0,00.html
Commission on Crystallographic Teaching Teaching Pamphlets : http://www.iucr.org/-->
Teaching Pamphlets or http://www.iucr.ac.uk-->
Teaching Pamphlets - International Union for Crystallography
Recommended articles to download:
1. A non-mathematical introduction to X-ray diffraction by C. A.
Taylor
2. An introduction to the scope, potential and applications of X-ray
analysis by M. Laing
6. Pourquoi les groupes de Symétrie en Cristallographie by
D. Weigel
9. Rotation Matrices and Translation Vectors in Crystallography
by S. Hovmöller
10. Metric Tensor and Symmetry Operations in Crystallography by
G. Rigault
11. The Stereographic Projection by E. J. W. Whittaker
13. Symmetry by L. S. Dent Glasser
14. Space Group Patterns by W. M. Meier
15. Elementary X-Ray Diffraction for Biologists by Jenny P. Glusker
21. Crystal Packing by Angelo Gavezzotti (download
here! - www download does not work)
22. Matrices, Mappings and Crystallographic Symmetry by Hans Wondratschek
Lecture Notes on QSAR by H. Kubinyi: http://home.t-online.de/home/kubinyi/
Materiais especiais para fazer download
Conformation of alkanes and cycloalkanes: Chapter
3 from F. A. Carey: Organic Chemistry, 4th ed., McGraw-Hill Co., NY, 2000.
Software para fazer download
Se usar qualquer programa ou banco de dados, deve
ser citado (o programa ou banco).
--------------------------------------------------------------------------------------------------------------------------------------------------------------------
Sofware para visualização
de estruturas moleculares / cristalográficas (gráficos moleculares):
Raswin para Windows ou Rasmol para outros sistemas:
http://www.rcsb.org/pdb/help-graphics.html
-->
Rasmol ou diretamente http://www.bernstein-plus-sons.com/software/rasmol/
Mercury para Windows ou outros sistemas: http://www.ccdc.cam.ac.uk/prods/mercury/index.html
-->
Mercury Download --> etc.
Download: http://www.ccdc.cam.ac.uk/prods/mercury/1_1_2/mercury_download_js.html
Documentação (manual): http://www.ccdc.cam.ac.uk/prods/mercury/docs/mercurydocn.pdf
DS ViewerPro 5.0, demo (30 days trial), Accelrys Inc.: http://www.accelrys.com/dstudio/ds_viewer/
Informação básica
/ Basic information


VMD
- Visual Molecular Dynamics, University of Illinois at Urbana-Champaign,
IL, USA: http://www.ks.uiuc.edu/Research/vmd/
--------------------------------------------------------------------------------------------------------------------------------------------------------------------
Sofware para análise de
estruturas moleculares / cristalográficas (análise estrutural):
PLATON for Windown v. 2000, download: files1
e / and files2
Instruções detalhadas / Detailed instructions, University
of Utrecht, The Netherlands: http://www.cryst.chem.uu.nl/platon/
--------------------------------------------------------------------------------------------------------------------------------------------------------------------
Software para cálculo de
propriedades moleculares

Dragon Web 3.0,
University of Milan, Italy: http://www.disat.unimib.it/chm/Dragon.htm
Input em SMILES format e alguns outros formatos.
O documento compactado contem a apresentação de Dragon
em ppt.
Lista de descritores moleculares:
list
Software para utilizar online
--------------------------------------------------------------------------------------------------------------------------------------------------------------------
LogP cálculos usando SMILES formato
de moléculas:
ALOGPS 2.1, Virtual
Computational Chemistry Laboratory: http://146.107.217.178/lab/alogps/-->
start the program



Molinspiration,
SMILES builder: http://www.molinspiration.com/services/logp.html
--------------------------------------------------------------------------------------------------------------------------------------------------------------------
Sofware para similarity search:



Molinspiration,
SMILES builder: http://www.molinspiration.com/services/index.html -->
Substructure, similarity and pharmacophore similarity searches
--------------------------------------------------------------------------------------------------------------------------------------------------------------------
Sofware para otimização
de geometria molecular (métodos químico-quânticos):

Hückel
MO, MOPAC, Gaussian, Gamess, University of Tasmania, Australia: http://www.chem.utas.edu.au/staff/yatesb/honours/modules/faces.html
-->
--> Hückel interfaces
--> MOPAC interfaces
--> Gaussian interfaces
--> Gamess interfaces
Um exemplo e input e output para o MOPAC:
http://www.chem.utas.edu.au/staff/yatesb/honours/modules/mod3/m_input.shtml
MOPAC 6.0 para Windows: program
e manual
Input somente com coordenadas internas
--------------------------------------------------------------------------------------------------------------------------------------------------------------------
Software para análise quimiométrica
/ Software for chemometric analysis

The
QSAR server, The University of North Carline, Chapel Hill, USA:
http://mmlin1.pha.unc.edu/~jin/QSAR/
Bancos de dados estruturais


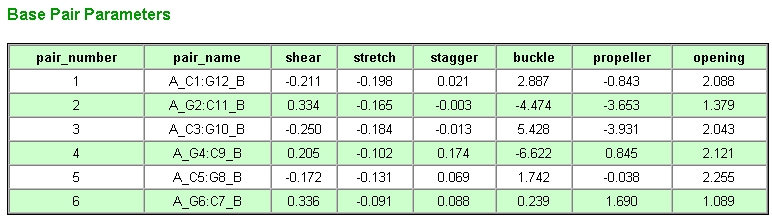
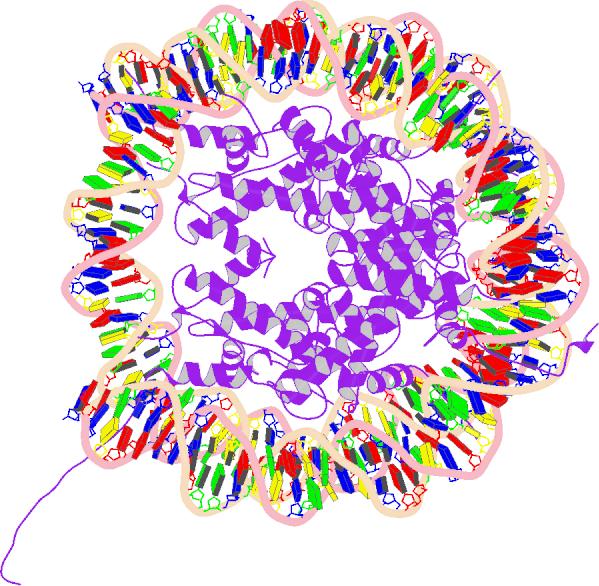
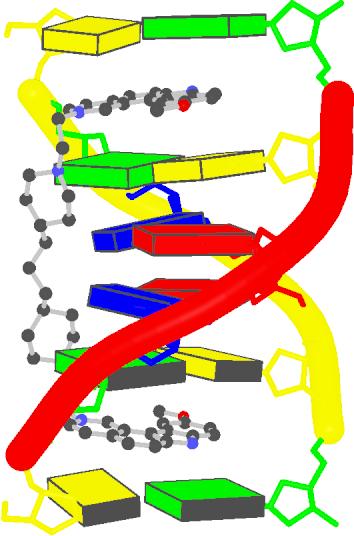
The
Nucleic Acid Database, The Rutgers University, New Jersey, USA:
http://ndbserver.rutgers.edu/NDB/index.html

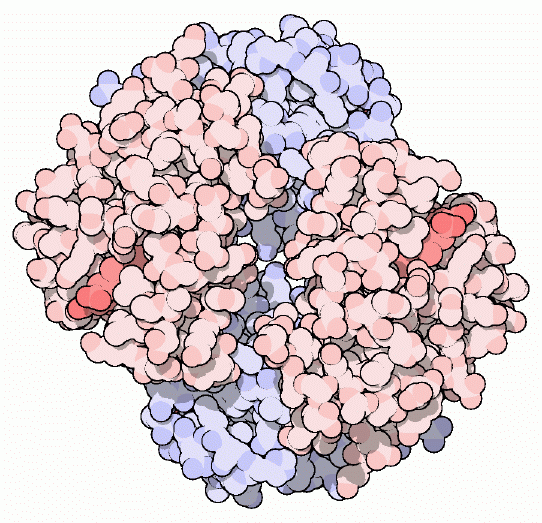

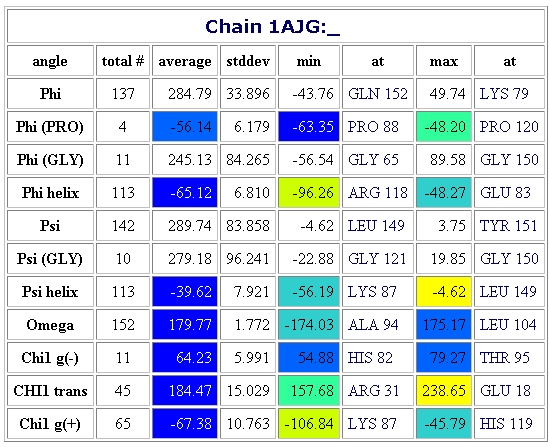
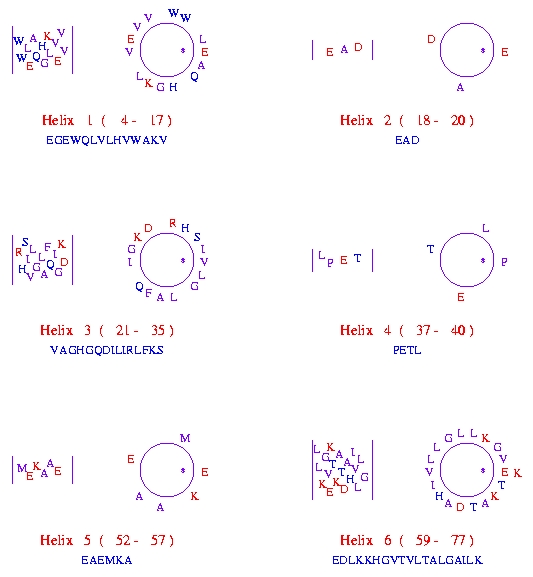
The Protein Data
Bank, The Rutgers University, New Jersey, USA: http://www.rcsb.org/pdb/
Mirror site,
Universidade Federal de Minas Gerais (UFMG), Belo Horizonte, MG, Brasil:
http://www.pdb.ufmg.br/pdb/
Tem várias ferramentas para análise/visualização
de biomacromoléculas e links para outros bancos de dados.
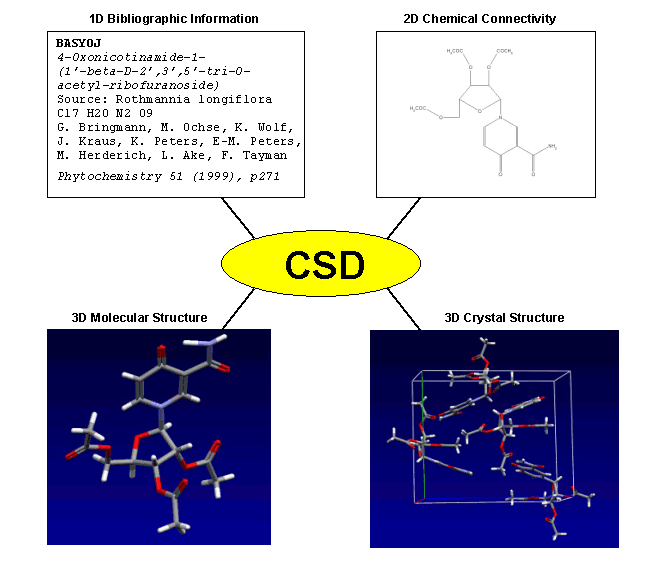

The
Cambridge Structural Database at NCSA Chem Viz, Univ. Urbana-Champaign,
Illinois, USA: http://chemviz.ncsa.uiuc.edu/content/homenew.html
--> Tools --> CSD
ou diretamente / or directly http://chemviz.ncsa.uiuc.edu/content/run-csd.html
É preciso se cadastrar para obter a conta e senha:
http://chemviz.ncsa.uiuc.edu/content/request_account.html
Informações básicas deste
banco de dados na página de CCDC, Cambridge Crystallographic Data
Centre, UK: http://www.ccdc.cam.ac.uk/
Se usar ester banco de dados, deve ser citado
(o banco) bem como o proprietário (NCSA Chem Viz).

NIST
Chemistry WebBook at NIST, USA: http://webbook.nist.gov/chemistry/
--> Search Options ou diretamente
http://webbook.nist.gov/chemistry/#Search
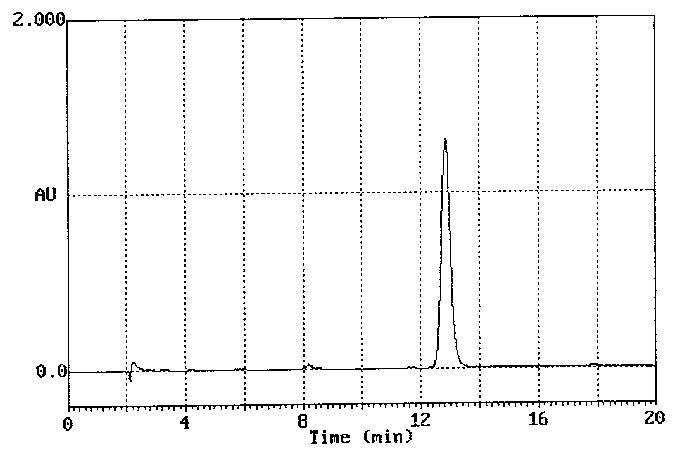
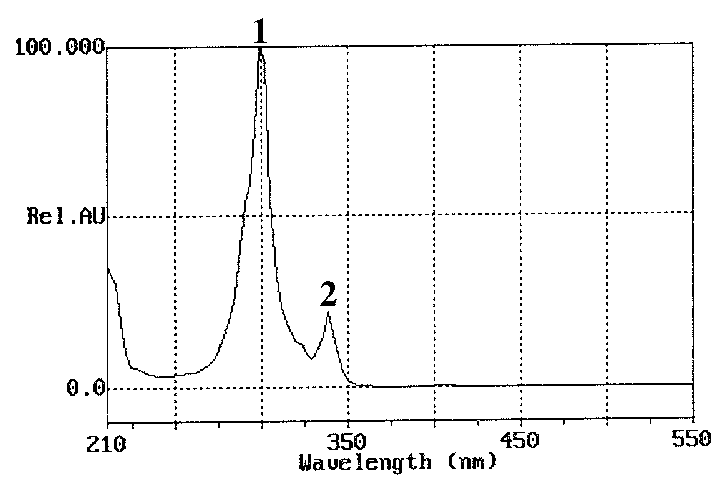
Polycyclic
Aromatic Hydrocarbons (PAHs) Database, Toyohashi University
of Technology, Japan: http://chrom.tutms.tut.ac.jp/JINNO/DATABASE/00database.html
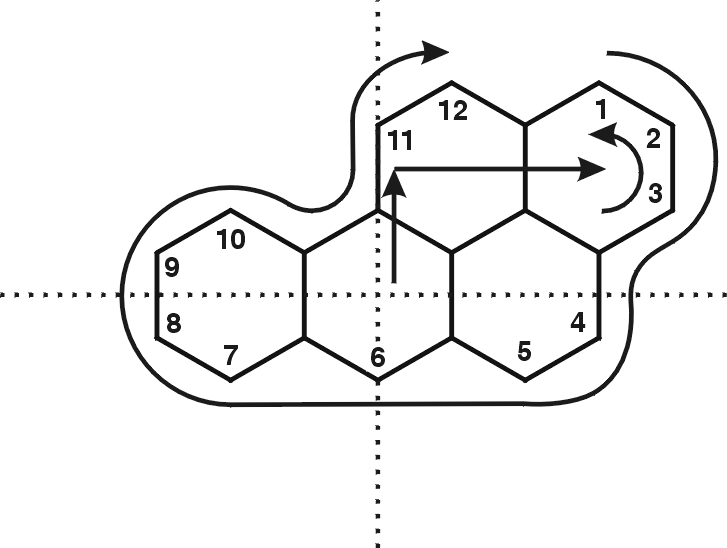
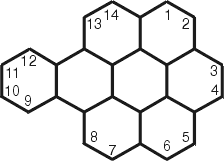

Polycyclic Aromatic
Hydrocarbon Structure Index, NIST, USA: http://ois.nist.gov/pah/
Introdução / Introduction



WebElements.com
at University of Sheffield, UK: http://www.webelements.com

3D
Molecular Modeling Home Page, Tokyo University of Pharmacy and
Life Science: http://triton.ps.toyaku.ac.jp/~dobashi/database/indexe.html


The IMB
Jena Image Library of Biological Macromolecules, University
of Jena, Germany: http://www.imb-jena.de/IMAGE.html
Tem várias ferramentas para análise/visualização
de biomacromoléculas e links para outros bancos de dados.

The Metalloprotein
Database and Browser, the Scripps Research Institute, La Jolla,
CA, USA: http://metallo.scripps.edu/


PROLYSIS
- a protease and protease inhibitor Web server, University of
Tours, France: http://delphi.phys.univ-tours.fr/Prolysis/


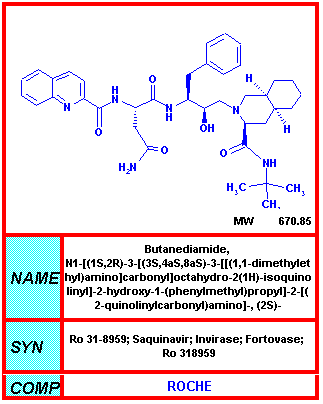
Database
for Anti-HIV Compounds, NIAID-NIST, USA: http://www.niaid.nih.gov/daids/dtpdb/protinh.htm
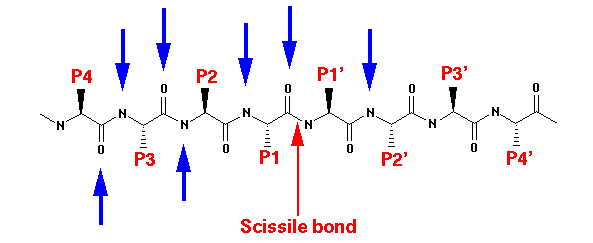
Proteinase
Inhibitor Database, the University of York, UK : http://www.ysbl.york.ac.uk/~proteinase/

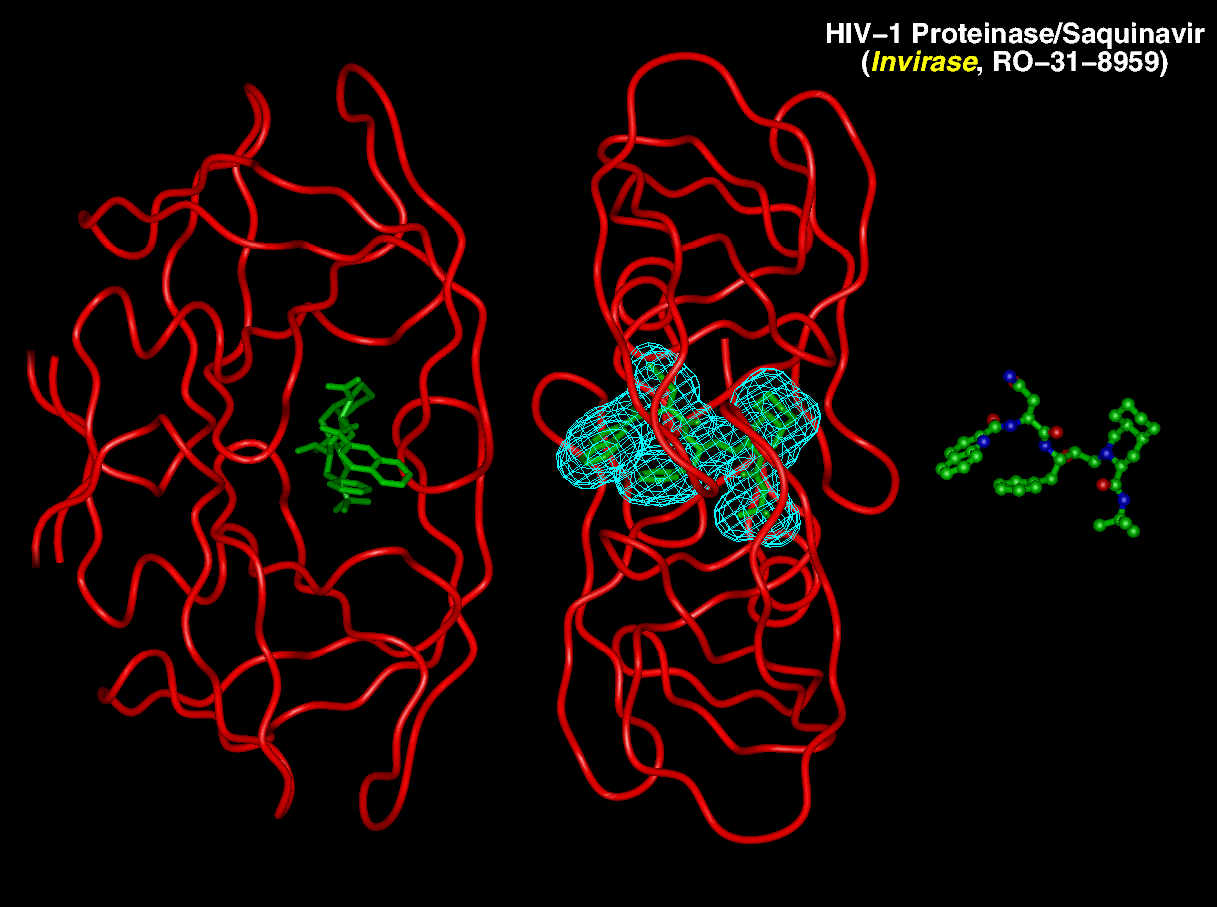
HIV
Protein Database, National Cancer Institute, USA: http://mcl1.ncifcrf.gov/hivdb/index.html
Links para vários bancos de dados estruturais:
http://xray.bmc.uu.se/embo/structdb/links.html
http://www.bio.unizh.ch/db/
http://www.xtal.iqfr.csic.es/ing/assistance.html
http://www.kisac.ki.se/education/courses/kth03/strdbs.html
http://www.life.uiuc.edu/z-huang/structldbs.html
http://swarna.ncsa.uiuc.edu/structdbs.html
http://www.rcsb.org/databases.html
http://www3.oup.co.uk/nar/database/cat/11
http://www.biochem.ucl.ac.uk/~robert/bioinf/lecture2/
etc.
Outros bancos de dados
Organic
Chemistry Database, Colby College, Waterville, Maine, USA :
http://www.colby.edu/chemistry/cmp/cmp.html

Klotho
- Biochemical Compounds Declarative Database: http://www.biocheminfo.org/klotho/

Development Therapeutics
Program, NCI-NIH, USA: http://dtp.nci.nih.gov/
Bancos de dados com atividades biológicas

Integrated
Spectral Data Base System for Organic Compounds, AIST, Japan:
http://www.aist.go.jp/RIODB/SDBS/sdbs/owa/sdbs_sea.cre_frame_sea
<--
Voltar para à versão portuguesa