Barbosa E. G., Martins J. P. A., Ferreira M. M. C., "LQTAgrid all force field, um software livre para o cálculo de descritores de campos moleculares para QSAR-4D" ["LQTAgrid all force field, a free sotware for calculation of molecular field descriptors in 4D-QSAR"]. Poços de Caldas, SP, 18-21/10/2009: XV Simpósio Brasileiro de Química Teórica [15th Brazilian Symposium of Theoretical Chemistry], Resumos [Abstracts] (2009) 335. Poster.
XV Simpósio Brasileiro de Química Teórica
C-II
LQTAgrid all force field, um software livre para o cálculo de descritores de campos moleculares para QSAR-4D
Euzébio Guimarães Barbosa (PG), João Paulo Ataide Martins (PG), Márcia Miguel Castro Ferreira (PQ)
Instituto de Química,
Universidade
Estadual de Campinas, CEP 13083-970 Campinas - SP.
Introdução
e objetivos
No formalismo CoMFA1, os descritores de campo
ou propriedades tridimensionais são determinados em uma caixa 3D
virtual ou grade (grid) que abrange todas as estruturas moleculares
alinhadas. Hopfinger et al.2 desenvolveram
uma metodologia independente de receptores (IR) que usa a
dinâmica molecular (DM) para incluir a dependência temporal
(4a
dimensão) em modelos de QSAR-3D. Com o intuito de combinar as
vantagens de ambos os métodos (CoMFA e QSAR-4D), foi
desenvolvida uma nova tecnologia chamada de LQTA-QSAR3
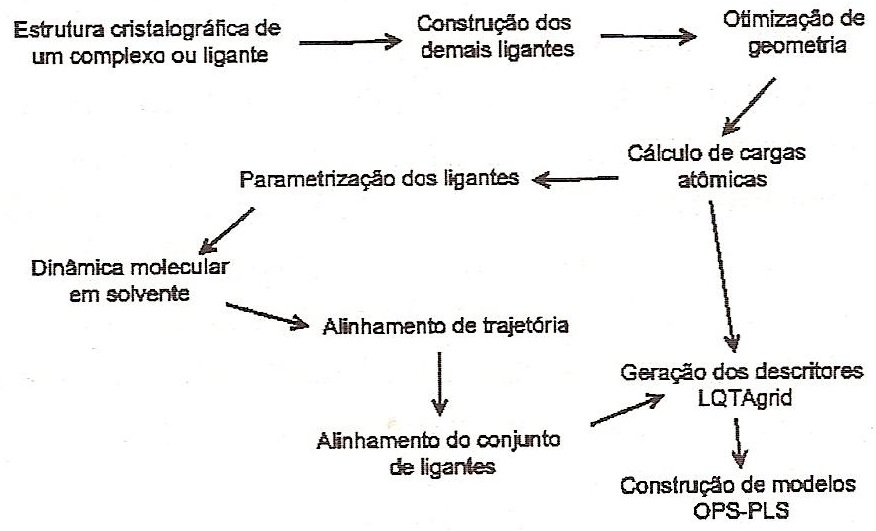
(Esquema 1).
Esquema 1. Passes necessários para uma análise LQTA-QSAR
Assim como acontece na
metodologia CoMFA, as moléculas são alinhadas em um grid
que é percorrido por uma ou mais sondas, que podem ser de
naturezas diferentes. Durante tal processo, são calculadas as
interações eletrostáticas e de van der Waals entre
a sonda e os átomos das moléculas do conjunto
investigado. No entanto, no LQTAgrid são consideradas as
interações entre a sonda e os átomos de todas as
conformações de cada molécula do conjunto
investigado, geradas em simulações de DM.
A primeira versão do programa LQTAgrid usa os parâmetros
de Lennard-Jones (LJ) derivados do campo de força ff43a1, nativo
do pacote GROMACS. Com o objetivo de estender o reconhecimento de novos
tipos de átomos foi desenvolvido o programa LQTAgrid all force field (LQTAgridAFF), no
qual o usuário tem a liberdade de definir os parâmetros de
LJ que julgar mais adequado para um determinado estudo utilizando a
metodologia LQTA-QSAR.
Métodos e
resultados
Para testar as novas potencialidades do programa LQTAgridAFF, foi
escholido o campo de força Generalized amber force field (GAFF),
pois ele é bastante adequado para o estudo de moléculas
orgânicas, tais como os compostos bioativos de estudos QSAR.
Primeiramente foram parametrizados ligantes com GAFF utilizando
ferramentas análogas ao programa Antechamber4-5
para a parametrização e criação de arquivos
de entrada adequados ao programa GROMACS. As trajetórias de
simulação foram alinhadas e usadas como entrada para o
programa LQTAgridAFF.
Conclusões
Resultados preliminares mostraram que o campo de força GAFF pode
ser usado em simulações de dinâmica molecular com o
GROMACS. O programa LQTAgridAFF, utilizando os parâmetros de LJ
importados do GAFF, gerou com sucesso descritores para a metodologia
LQTA-QSAR. O programa em breve estará disponível no site
lqta.iqm.unicamp.br.
Agradecimentos
CAPES, CNPq e FAPESP.
335
English
XV Simpósio Brasileiro de Química Teórica
C-II
LQTAgrid all force field, a free software for calculation of molecular field descriptors in QSAR-4D
Euzébio Guimarães Barbosa (PG), João Paulo Ataide Martins (PG), Márcia Miguel Castro Ferreira (PQ)
Instituto de Química,
Universidade
Estadual de Campinas, CEP 13083-970 Campinas - SP.
Introduction and
goals
Molecular field descriptors or three-dimensional properties in the
CoMFA formalism1 are calculated in a virtual
3D box or grid inside which all molecular structures are aligned.
Hopfinger et al.2 have developed the
receptor-independent (IR) methodology which uses molecular dynamics
(MD) to include the temporal dependence
(the 4th
dimension) in QSAR-3D models. A new technology named LQTA-QSAR3
(Scheme 1)
has been developed as the combination of advantages of the both methods
(CoMFA and QSAR-4D).
Scheme 1. Steps in a LQTA-QSAR analysis
Similarly as in the
CoMFA methodology, molecules are aligned in a grid in which one or more
probes of different nature are used. During this procedure,
electrostatic and van der Waals energies are calculated for
interactions between atoms of the probe and the molecular set. However,
LQTAgrid considers interactions between a probe and atoms of all
conformations of each molecule from the molecular set as determined in
MD simulations.
The first version of program LQTAgrid uses Lennard-Jones (LJ)
parameters of the ff43a1 force field from the GROMACS package. Program LQTAgrid all force field (LQTAgridAFF) was
developed with the purpose to extend the force filed to new atom types,
what enables to user to define freely LJ parameters whenever necessary
in a study that is based on the LQTA-QSAR methodology.
Methods and
results
A new force field called Generalized amber force field (GAFF) was selected to test
the capabilities of program LQTAgridAFF, because this force field is suitable for
treatment of organic molecules such as bioactive compounds in QSAR
studies.
As the first step, ligands were parameterized by GAFF using tools
similar to those of program Antechamber4-5
for parameterization and generation of input files for GROMACS.
Simulation trajectories were aligned and then used as inputs for
program LQTAgridAFF.
Conclusions
Preliminary results have shown that the GAFF force field can be used in
GROMACS molecular dynamics simulations. The program LQTAgridAFF which
used LJ parameters from GAFF worked satisfactory in generating
descriptors for the
LQTA-QSAR methodology. The program will be soon available online on
site
lqta.iqm.unicamp.br.
Acknowledgements
CAPES, CNPq and FAPESP.
335